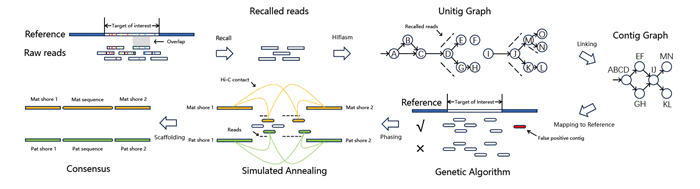
近日,中國農業科學院農業基因組研究所農業基因組學技術研發與應用創新團隊開發出了一種新算法——TRFill,解決了現有工具無法完全填補基因組間隙的難題,顯著提升了基因組質量。相關研究成果發表在《基因組生物學(Genome Biology)》上。
動植物基因組的許多區域存在大量高度重復的DNA片段(如人類著絲粒、番茄端粒附近區域),這些重復結構會干擾基因組的準確拼接,導致測序結果出現錯誤或缺失。由于重復區域的復雜性,現有的組裝軟件難以完全填補基因組間隙。
為此,研究團隊開發了一種名為TRFill的新算法,該算法能有效解決動植物基因組測序中的重復序列難題。TRFill利用兩種高精度測序數據(PacBio HiFi長讀序列和Hi-C染色體空間信息),成功填補了基因組組裝中的重復區域缺口。經測試,在人類著絲粒和番茄端粒這些傳統測序難題區域,該算法能夠成功修復近三分之二的重復序列結構。特別在番茄中,完善的端粒重復序列圖譜首次讓研究人員能夠從群體層面分析這些復雜結構的變異規律。該技術為繪制更完整的基因組圖譜、研究重復序列功能提供了重要工具。
該研究得到國家自然科學基金、中國農業科學院科技創新工程等項目資助。(通訊員 馬昕怡)
原文鏈接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03685-5